Le génotypage couplé au phénotypage permet de caractériser plus rapidement les variétés.
Rapidité, essais raisonnés, maîtrise des coûts... Combiner le marquage moléculaire à du phénotypage classique accélère le processus de caractérisation des variétés de blé tendre et d'orge à l'inscription.
Débuté en 2020, le projet Casdar Cap-Phenogen (2020-2023), coordonné par le Geves en partenariat avec Arvalis, Inrae et l'UFS, a établi la faisabilité d'une nouvelle méthode pour caractériser les résistances variétales pour le blé tendre et l'orge dans le dispositif d'inscription au Catalogue français dès leur première année d'évaluation.
Le marquage moléculaire, qu'est-ce que c'est ?
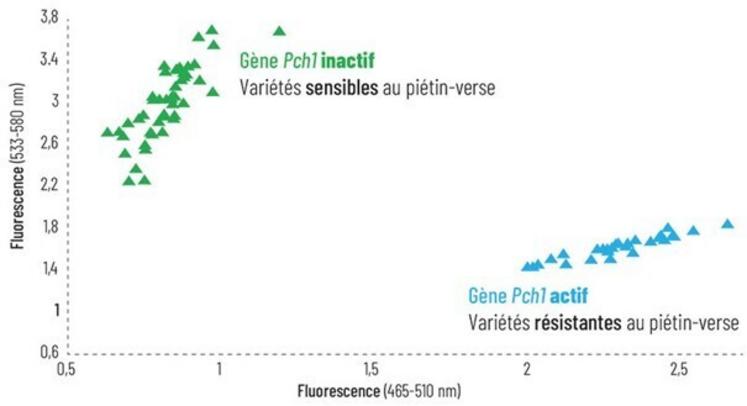
Un marqueur moléculaire est une sorte " d'étiquette " spécifique à une région précise de l'ADN ou à un gène cible. Il met littéralement en lumière la présence ou l'absence d'un gène, ou encore les différentes formes actives et inactives d'un gène, par des techniques de laboratoire telle que la fluorescence (figure 1).
Le marquage moléculaire permet d'identifier la présence ou l'absence, chez les variétés à l'inscription, d'un gène de résistance à une maladie donnée (sur la figure 1 le piétin-verse) en 2 heures, contre plusieurs mois par la méthode traditionnelle. Chaque point correspond à une variété testée, dûment répertoriée.
Le génotypage par marquage moléculaire consiste donc à utiliser un marqueur moléculaire pour suivre une région de l'ADN ou un gène d'intérêt agronomique dans les variétés cultivées, et prédire ainsi leur comportement potentiel avant même leur évaluation au champ.
Le projet Cap-Phenogen s'est concentré sur six combinaisons "Céréale/Pathogène/Gène-cible" : trois pour le blé tendre et trois pour l'orge. Réalisé en laboratoire, ce marquage moléculaire peut être utilisé dès la première année d'étude des variétés arrivant dans le dispositif VATE. Il repère ainsi immédiatement les nouvelles variétés qui possèdent le gène de résistance à la maladie étudiée.
Moins de variétés à tester en deuxième année d'inscription
Le tri en année 1 réalisé par la nouvelle méthode réduit drastiquement le nombre d'essais ou de variétés à implanter au champ en 2e année d'évaluation. Une variété dont le phénotype au champ présente une résistance en année 1 et dont la forme résistante du gène associé a été identifiée par marquage moléculaire, sera jugée résistante dès la première année.
En deuxième année d'évaluation VATE, les essais se focalisent seulement sur les variétés où il y a une incertitude sur le phénotypage ou un désaccord entre le marquage moléculaire et le phénotypage au champ, pour une validation du phénotypage au champ.
Des règles de décision CTPS ont été établies pour chaque combinaison " Hôte/Maladie/Gène-cible ", où le phénotypage au champ prévaut sur le marquage. Les marqueurs moléculaires établissent la présence de quelques gènes majeurs de résistance, faciles à suivre.
L'absence d'un gène ne signifie pas forcément "sensibilité" à la maladie, car la variété peut avoir d'autres gènes de résistance qu'on ne connaît pas encore pour le moment.
À l'inverse, la présence d'un gène ne signifie pas forcément de la résistance en condition de culture, du fait de l'environnement pédoclimatique qui peut influencer l'expression des caractères génétiques, ou du contournement du gène par l'apparition d'un nouveau variant du pathogène.
C'est pourquoi la nouvelle méthode continue d'inclure l'habituel phénotypage au champ de l'ensemble des variétés à l'inscription, afin de confirmer la résistance repérée par marquage moléculaire.
Les variétés dont le phénotypage au champ révèle une résistance non liée au marqueur pourront, après expertise, faire une deuxième année de phénotypage pour confirmer cette résistance. " Grâce à cette méthode combinant génotypage et phénotypage, nous avons eu en 2024 une réduction d'implantation de 96 % pour le piétin-verse et de 94 % pour les mosaïques en blé tendre en année 2 ", se réjouit Valérie Cadot (Geves), coordinatrice du projet.